目录
Bulk Si system (Active Leanring)
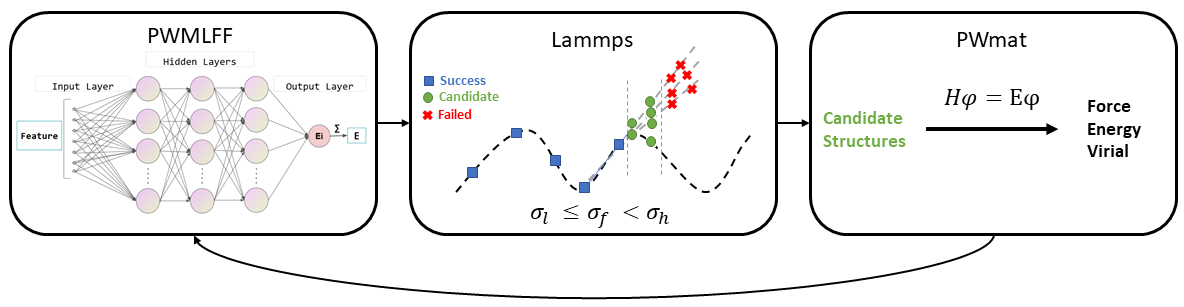
下文将以 Bulk Si 系统为例,介绍如何使用 PWMLFF Deep Potential Model 进行训练,同时结合主动学习加强模型泛化性,并进行 lammps 模拟。
力场训练结束时得到的力场文件为*.ff,该文件包含了训练得到的所有参数,可用于 lammps 模拟。模拟时发现模型在同一个体系,不同温度下的表现是不同的,因此需要针对不同温度下的体系进行训练。在本例中,我们首先测试模型在 3 个不同温度下的表现,分别为 300K、500K、900K。根据下述教程可以发现,模型在 900K 下的表现非常差,因此我们需要针对 900K 下的体系进行训练,以提高模型的泛化性,准确描述该体系在 300K - 900K 区间的动力学行为。
主动学习流程如下图所示:
主动学习工作目录整体结构示意
Si_system/adaptive_trainer
└── dir
├── 00.initdata # 初始数据集(AIMD)
│ ├── perturb.py # 微扰结构,执行文件
│ └── Structure ( Si64 ) # 初始结构 'atom.config' 存放目录,以Si64为例
│ ├── atom.config # 初始结构,用于产生数据集
│ ├── structures # 执行perturb.py后产生的结构所存放的目录
│ └── AIMD # AIMD(初始数据集)结果存放目录
│ ├──md-0 # 从structures中提取的结构文件存放目录
│ └──md-..
│
├── 01.Iteration1 # 初次迭代目录
│ ├── 00.train # 初始训练集(generate_data),必须为00.train
│ │ ├── forcefield_0 # 4个力场训练(load_and_train & extract_force_field)
│ │ ├── forcefield_1 # 可以训练多个力场
│ │ ├── forcefield_2 # 每个目录下包含一个力场文件
│ │ └── forcefield_3
│ ├── 01.explore # 探索目录
│ │ ├── explore
│ │ ├── model # 存放力场文件的目录
│ │ ├── result # 存放lammps计算后筛选出的结构文件的目录
│ │ └── subsys # 从Strucutre中获取结构文件,并进行lammps计算
│ └── etot.input # 用于完成PWmat自洽计算
│
├── 02.Iteration2 # 第二次迭代目录...再原有数据集基础上添加新的训练集再次进行训练及探索
│ ├── 00.train
│ └── 01.explore
│
│
├── 03.Iteration3 # 第三次迭代目录...
│ ├── 00.train
│ └── 01.explore
│
└── 04.Iteration4 # 第四次迭代目录...
├── 00.train
└── 01.explore
1. 初始数据准备
为了获取相同体系下不同的初始结构,用于进行 AIMD 计算,获取原子运动轨迹。在本例中,我们提供了一个方式,将使用perturb.py对原子进行晶格及原子位置微扰,生成 50 个初始结构,用于进行 AIMD 计算。perturb.py的使用方法如下:
主动学习模块引入了新的接口,需要提前在 PWMLFF package 中安装:
在线安装:
$ conda activate PWMLFF $ cd path_to/PWMLFF/src $ git clone https://github.com/lhycms/MaterSDK.git $ cd MaterSDK $ pip install .
源码:
https://github.com/lhycms/MaterSDK
新建Si64目录,将并在目录下存放初始结构atom.config。准备完成后直接执行python perturb.py即可,执行后会在Si64目录下生成structures目录,其中存放了扰动后的结构文件,同时在Si64目录下生成AIMD目录,其中同样存放了扰动后的结构文件,之后用于 AIMD 计算获取原子运动轨迹。
以下是perturb.py的示例:
from MaterSDK.matersdk.adalearn.generator.perturbation import BatchPerturbStructure import os if __name__ == "__main__": """ 1. only need to run once!!! 2. perturb the structure 3. seed for adaptive sampling """ Perturbed = ['Si64'] pert_num = 50 cell_pert_fraction = 0.03 atom_pert_distance = 0.01 BatchPerturbStructure.batch_perturb( Perturbed=Perturbed, pert_num=pert_num, cell_pert_fraction=cell_pert_fraction, atom_pert_distance=atom_pert_distance, ) aimd_directory = os.path.join(os.path.abspath(Perturbed[0]), 'AIMD') if not os.path.exists(aimd_directory): os.makedirs(aimd_directory) # Create 'md-0', 'md-1', ..., 'md49' directories under 'AIMD' directory for i in range(pert_num): md_directory = os.path.join(aimd_directory, f'md-{i}') if not os.path.exists(md_directory): os.makedirs(md_directory) # Link the corresponding config file from 'structures' directory to 'md-{i}' directory config_file = os.path.join(os.path.abspath(Perturbed[0]), 'structures', f'{i}.config') link_file = os.path.join(md_directory, 'atom.config') if os.path.islink(link_file): os.remove(link_file) os.symlink(config_file, link_file)
Perturbed需要指定所存放的atom.config所在目录的名称pert_num为每个体系的扰动结构数目,即需要根据提供的atom.config对结构进行包括晶格和原子位置的扰动,以增强结构的采样变化cell_pert_fraction为扰动的晶胞尺寸atom_pert_distance为扰动的原子位置
进入AIMD目录,准备一个etot.input文件,内容示例如下,用户可以根据具体的体系完成设置,参数意义参考PWmat manual:
4 1 job = MD MD_DETAIL = 2 10 1 1000 1000 XCFUNCTIONAL = PBE in.atom = atom.config mp_n123 = 2 2 2 0 0 0 3 ecut = 60 ecut2 = 240 ENERGY_DECOMP = T OUT.STRESS = F in.psp1 = Si.SG15.PBE.UPF
- 可选项
ENERGY_DECOMP:是否将总 DFT 能量分解为属于每个原子的能量(原子能量)。结果输出在MOVEMENT文件中。如需使用或训练原子能量,需要将其设置为T。 - 可选项
OUT.STRESS:是否输出应力信息,如需训练Virial,则需要将其设置为T。
- 这里仅使用 10fs 的时间步长,实际使用中需要根据体系进行设置。50 个结构拼接起来的
MOVEMENT轨迹有 500 帧,即 500fs. - 可以使用以下命令将 PWmat 输入文件
etot.input及 slurm 作业脚本slurm.sh拷贝到md-0, md-1, ..., md-49目录下:
for i in {0..49} do cd md-$i ln -s ../etot.input ln -s ../slurm.sh ln -s ../Si.SG15.PBE.UPF sbatch slurm.sh cd .. done
- 计算完成后,可以使用以下命令将所有结构的
MOVEMENT拼接起来:
for i in {0..49} do cat md-$i/MOVEMENT >> MOVEMENT done
该MOVEMENT文件将用于后续的训练。
2. 初始训练集
将上述AIMD获取的MOVEMENT文件放置于00.train目录下,新建*.json文件(如dp.json),该文件包含一系列需要传入的参数。(详情见输入文件部分)
2.1 输入文件
以下是训练时的参数设置:
{
"recover_train":false,
"work_dir":"work_train_dir",
"reserve_work_dir": false,
"train_movement_file":["MOVEMENT"],
"forcefield_name": "forcefield.ff",
"forcefield_dir": "forcefield",
"train_valid_ratio":0.8,
"seed":712350,
"model_type": "DP",
"model_num" : 4,
"atom_type":[14],
"max_neigh_num":100,
"model":{
"descriptor": {
"Rmax":6.0,
"Rmin":0.5,
"M2":16,
"network_size":[25, 25, 25]
},
"fitting_net": {
"network_size": [50, 50, 50, 1]
}
},
"optimizer":{
"optimizer":"LKF",
"block_size":5120,
"kalman_lambda":0.98,
"kalman_nue":0.99870,
"nselect":24,
"groupsize":6,
"batch_size": 4,
"epochs":20,
"start_epoch":1,
"print_freq":10,
"train_energy":true,
"train_force":true,
"train_ei":false,
"train_virial":false,
"train_egroup":false,
"pre_fac_force":2.0,
"pre_fac_etot":1.0,
"pre_fac_ei":1.0,
"pre_fac_virial":1.0,
"pre_fac_egroup":0.1
}
}
- 多个模型的训练需要指定关键词
model_num。在本例中,我们将训练 4 个模型。 - 需要注意的是,对于 4 个模型下的训练,需要为不同的训练设置不同的种子数
seed。训练时使用的仍是相同的feature文件。
2.2 运行
- 准备一个
slurm脚本 (如slurm.sh) 用于提交多个训练。
以下 slurm 示例脚本头文件适用于 Mcloud,提交任务时确保已经加载必要的环境和模块。
#!/bin/sh #SBATCH --partition=3080ti #SBATCH --job-name=mlff #SBATCH --nodes=1 #SBATCH --ntasks-per-node=1 #SBATCH --gres=gpu:1 #SBATCH --gpus-per-task=1
1. 提交任务:
PWMLFF train dp.json slurm.sh > log
3. 主动学习探索过程
初始多个力场生成后,新建目录01.explore,并准备etot.input输入文件用于后续的自洽计算,以生成新的数据集用于二次训练。
- 训练完成后,请检查每个model目录下的
epoch_train.dat及epoch_valid.dat文件,确保训练 loss 达到一定的收敛程度。
[]./..(pictures/epoch-train&valid_dat4.png)
loss对应训练总误差RMSE_Etot_per_atom对应训练能量误差,建议达到 ~$10^{-3} eV/atom$ 数量级RMSE_F对应训练力误差, 建议达到 ~$10^{-2} eV/\text{\AA}$ 数量级- 如果训练集的误差比验证集的误差明显偏小,表明训练过拟合,可适当增加训练集的大小或调整 batch_size 的数量。
2. 训练完成后生成的*.ff力场文件需要用于 lammps 模拟,请下载经过修改的版本重新编译 lammps
其中etot.input文件内容示例如下,用户可以根据具体的体系完成设置,参数意义参考PWmat manual:
4 1 job = scf in.atom = atom.config in.psp1 = /share/psp/NCPP-SG15-PBE/Si.SG15.PBE.UPF accuracy = high ecut = 60.0 wg_error = 0.0 e_error = 0.0001 rho_error = 0.0 out.wg = F out.rho = F out.vr = F out.force = T out.stress = T out.mlmd = T energy_decomp = T
out.mlmd = T,out.force = T。- 请使用最新版本的 PWmat
- 该模板并未指定
mp_n123,将由后续的adaptive_trainer根据kspacing自动设置。
使用力场调用lammps进行探索时,输入文件内容示例如下,用户可以根据具体的体系完成设置。
{
"psp_dir" : "/share/psp/NCPP-SG15-PBE",
"struct_dir" : "/data/home/hfhuang/2_MLFF/2-DP/19-json-version/3-Si/00.initdata/Si64",
"model_type" : "DP",
"model_num" : 4,
"ff_file" : ["/data/home/hfhuang/2_MLFF/2-DP/19-json-version/3-Si/01.Iter1/00.train/forcefield_0/forcefield.ff",
"/data/home/hfhuang/2_MLFF/2-DP/19-json-version/3-Si/01.Iter1/00.train/forcefield_1/forcefield.ff",
"/data/home/hfhuang/2_MLFF/2-DP/19-json-version/3-Si/01.Iter1/00.train/forcefield_2/forcefield.ff",
"/data/home/hfhuang/2_MLFF/2-DP/19-json-version/3-Si/01.Iter1/00.train/forcefield_3/forcefield.ff"],
"etot_file" : "./etot.input",
"ensemble" : "nvt",
"temp" : [300,500,900],
"pressure" : [1.0],
"atom_type" : [14],
"md_dt" : 0.001,
"traj_step" : 100,
"process_num" : 64,
"lmp_nprocs" : 4,
"num_select_per_group" : 200,
"kspacing" : 0.16,
"success_bar" : 0.15,
"candidate_bar" : 0.35
}
psp_dir为所使用的赝势文件目录所在路径struct_dir为所使用的初始结构文件目录所在路径,对应前面的Perturbed目录。此时需要读取之前微扰产生的结构文件用于 lammps 计算,具体用途见下文model_type为所使用的力场类型model_num为所使用的力场数目ff_dir为所使用的力场文件etot_file为所使用的自洽计算输入文件所在路径ensemble为所使用的分子动力学系综,如nvt,npttemp为 lammps 计算的温度范围(lammps 输入文件参数设置)pressure为 lammps 计算的压力范围atom_type为所使用的原子类型,与前面训练时保持一致的排序md_dt为 lammps 的 md 步长,目前仅支持unit metal,0.001对应1fstraj_step为 lammps 的 md 步数,初次探索时可以设置为较小的值,如500,后续探索时根据 error 结果逐步增大,如1000,2000,4000等process_num为所使用的 lammps 计算总核数,需要保证process_num为lmp_nprocs的整数倍lmp_nprocs为每个 lammps 计算进程所使用的 cpu 核数num_select_per_group为最后从 lammps 计算中随机选取的结构数目kspacing为所使用的 k 点取值间隔,单位为$\frac{1}{\text{\AA}}$success_bar为所使用的探索成功率置信区间$\sigma_l$,初次探索时可以设置为较大的值,如0.15,后续探索时可以缩小范围,如0.12,0.1,0.08等candidate_bar为所使用的探索候选率置信区间$\sigma_h$,初次探索时可以设置为较大的值,如0.35,后续探索时可以缩小范围,如0.25,0.2,0.18等
- $\sigma_f < \sigma_l$ 表明区间内对应结构的原子受力都被准确地预测,不需要进行 DFT 计算。
- $\sigma_f \ge \sigma_h$ 表明势函数预测相应结构的原子受力误差太大,不适合作为升级势函数的候选结构。
- $\sigma_l \le \sigma_f < \sigma_h$ 表明势函数预测相应结构的原子受力有一定误差,又不会太大,最适合作为升级当前势函数的候选结构,将会从中随机抽样
num_select_per_group个结构进行 DFT 计算打上标签,获取新的数据集。 - 执行该
.py文件时,确保环境变量中存在PWmat主程序。
3.1 运行
以下 slurm 示例脚本适用于 Mcloud,提交任务时确保已经加载必要的环境和模块。如module load pwmat等。
#!/bin/sh #SBATCH --partition=cpu #SBATCH --job-name=lmp #SBATCH --nodes=1 #SBATCH --ntasks-per-node=64 echo "Starting job $SLURM_JOB_ID at " `date` echo "SLURM_SUBMIT_DIR is $SLURM_SUBMIT_DIR" echo "Running on nodes: $SLURM_NODELIST" PWMLFF explore explore.json echo "Job $SLURM_JOB_ID done at " `date`
- –ntasks-per-node=64 与
process_num的值一致。
程序运行结束后,可以检查当前目录下生成的explore.ratio文件,其内容如下:
num of all img: 73815 ratio of success: 0.8031294452347084 ratio of candidate: 0.19326695116168802 ratio of failure: 0.0036036036036036037
num of all img为探索过程中所有的结构数目(trajectory)ratio of success为探索成功率ratio of candidate为探索候选率,即筛选出可用于自洽计算的结构数目的比率ratio of failure为探索失败率
在./explore/result目录下生成num_select_per_group个结构文件及etot.input文件。
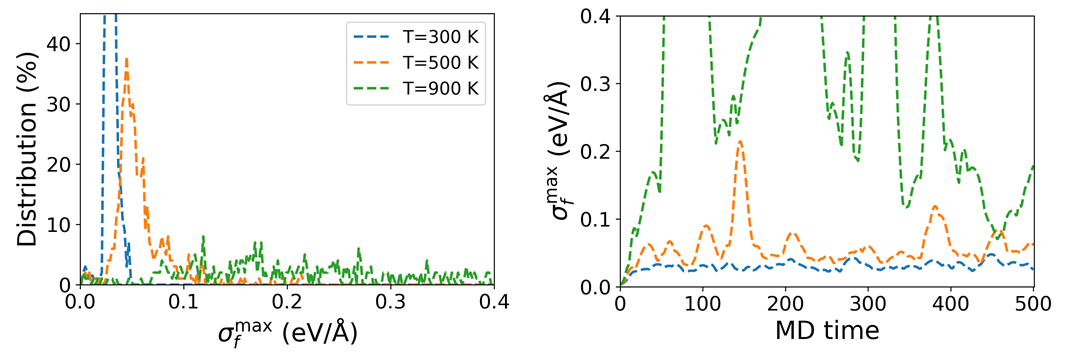
同时会生成nvt_explr_plot目录,其中dev_fmax.png文件,为不同温度/压力下探索成功率和探索候选率的置信区间分布。
如下图所示
根据探索时设置的success_bar和candidate_bar,可以确定探索成功率和探索候选率的置信区间。如图中 300K 和 500K 在分布在 0.1 以内的峰值(Distribution)越高,说明探索成功率和探索候选率越高,即对应力场的在 300K 和 500K 时训练效果表现越好。
而根据$\sigma_f^{max}$随 MD 步数变化的曲线,可以确定探索的收敛性。如图中 300K 的$\sigma_f^{max}$随 MD 步数变化的曲线趋向于平缓,说明探索已经收敛,即对应力场的在 300K 时训练效果表现越好。
4. 迭代探索
迭代探索的目的是为了进一步提高力场的泛用性,即在更多的温度/压力下,探索成功率和探索候选率都能达到较高的置信区间。后续教程中以 bulk Si 在 900K 时的径向分布函数来验证力场的性能。
具体做法是将初次探索时得到的存储在/explore/result目录下结构文件进行静态计算,并将所有结构静态计算得到的OUT.MLMD文件拼接成一个新的MOVEMENT文件。然后将其加入到数据集中(目录下包含原有的MOVEMENT),重新训练力场,再进行探索。
计算完成后,请检查所有计算是否收敛,若有未收敛的计算,请重新计算。以下是一个用于检查的示例脚本:
#!/bin/bash directories=($(find . -type f -name "etot.input" -exec dirname {} \;)) ############################## sum_n=0 sum_t=0 no_converge_directories=() scf_stop_directories=() for i in "${directories[@]}"; do niter_count=$(grep -c "niter" "$i/REPORT") time_count=$(grep -c "time" "$i/REPORT") sum_n=$((sum_n + niter_count)) sum_t=$((sum_t + time_count)) if [ "$niter_count" -eq 1 ]; then no_converge_directories+=("$i") fi if [ "$time_count" -eq 0 ]; then scf_stop_directories+=("$i") fi done echo "Number of scf failures: $sum_n" echo "Number of scf success: $sum_t" echo "No converge in: ${no_converge_directories[*]}" echo "scf stop in: ${scf_stop_directories[*]}"
$ bash test.sh Number of scf failures: 1 Number of scf success: 99 No converge in: ./12 scf stop in:
计算完成后,可使用以下命令将OUT.MLMD拼接为一个新的MOVEMENT:
for i in {0..199} do cat $i/OUT.MLMD >> MOVEMENT done
迭代探索完成后,同样根据dev_fmax.png图中$\sigma_f^{max}$的分布曲线,可以确定力场的表现。
结果如下图所示
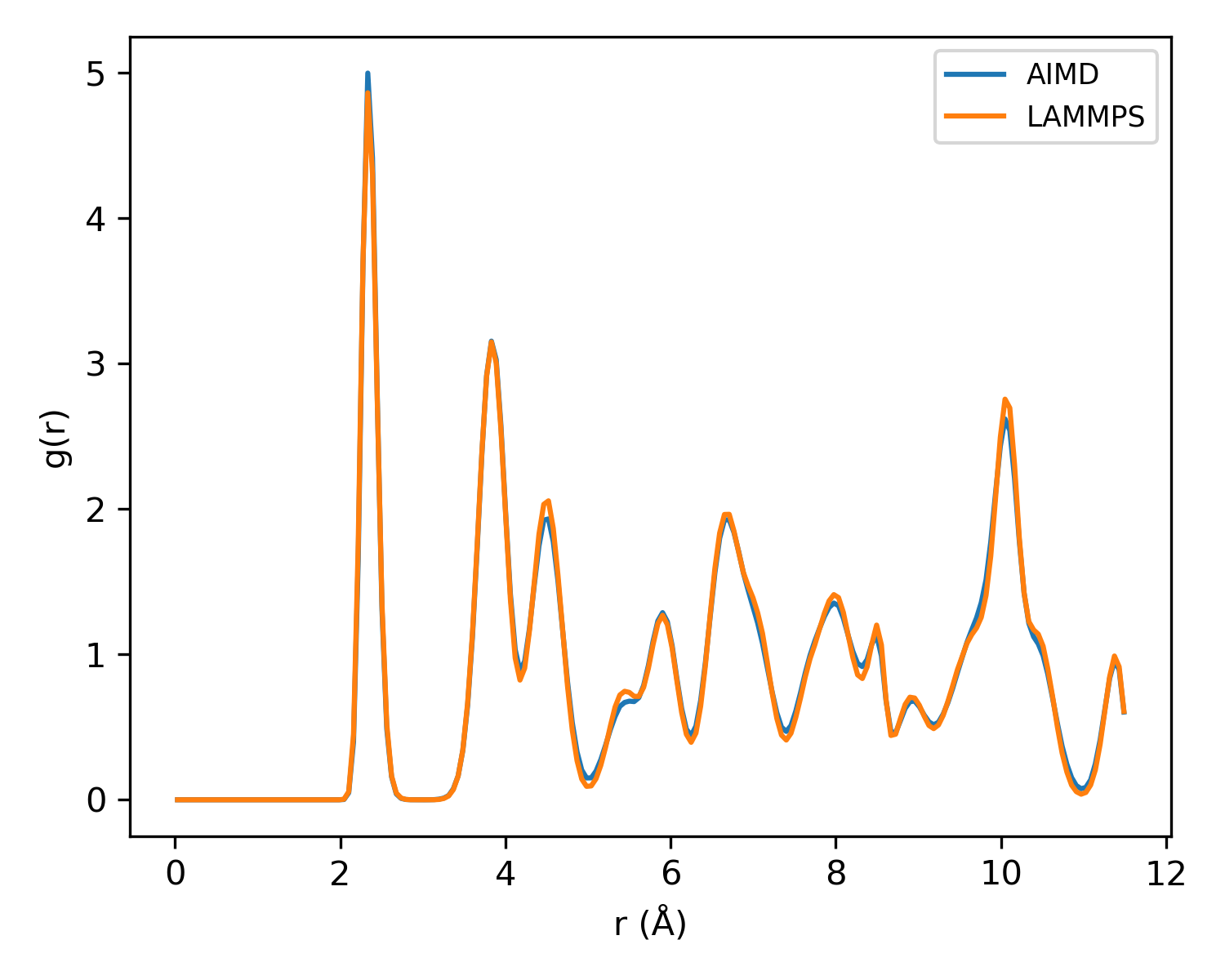
经过多次的迭代探索,探索成功率和探索候选率的置信区间都达到了较高的值。接着可以使用该力场进行 bulk Si (216 atoms)在 900K 时的径向分布函数的计算,结果如下图所示
可以看到,使用 bulk Si (64 atoms) 小结构进行训练得到的力场,却能正确描述 builk Si (216 atoms)大结构的动力学行为,即使用该力场进行 lammps 计算得到的径向分布函数与 DFT 计算得到的结果吻合较好。